陈庭坚教授团队在NAR发表论文:真核DNA聚合酶对非天然碱基对的识别及通用扩展遗传字母测序技术的建立
近日,华南理工大学生物科学与工程学院陈庭坚教授团队在真核生物DNA聚合酶对非天然碱基对的识别及其在非天然碱基DNA测序技术开发中的应用研究方面取得重要进展。团队首次系统探索并展示了人源DNA聚合酶β(Pol β)对多种非天然碱基对的识别能力,并基于此创造性地开发出一套普适、精准的非天然碱基DNA测序技术,成功实现了对包含单个乃至多个非天然碱基的DNA序列的读取。该成果又发表在国际顶级期刊Nucleic Acids Research上,题目为“Recognition of unnatural base pairs by a eukaryotic DNA polymerase enables universal sequencing of an expanded genetic alphabet”。硕士研究生罗汉涛、副研究员杜宇辉和博士后孙乐平为论文的共同第一作者,陈庭坚教授为唯一通讯作者,华南理工大学生物科学与工程学院为唯一完成单位。(论文链接:https://doi.org/10.1093/nar/gkaf1460)

生命体的遗传信息由A、T、C、G四种天然碱基编码。为突破天然遗传信息的编码极限,科学家致力于在DNA中引入可复制的非天然碱基对,以构建扩展的遗传字母表。然而,非天然碱基对能否被真核细胞自身的复制机器有效识别并复制,以及如何对含有非天然碱基的DNA进行高效测序,是两大长期存在的核心挑战。
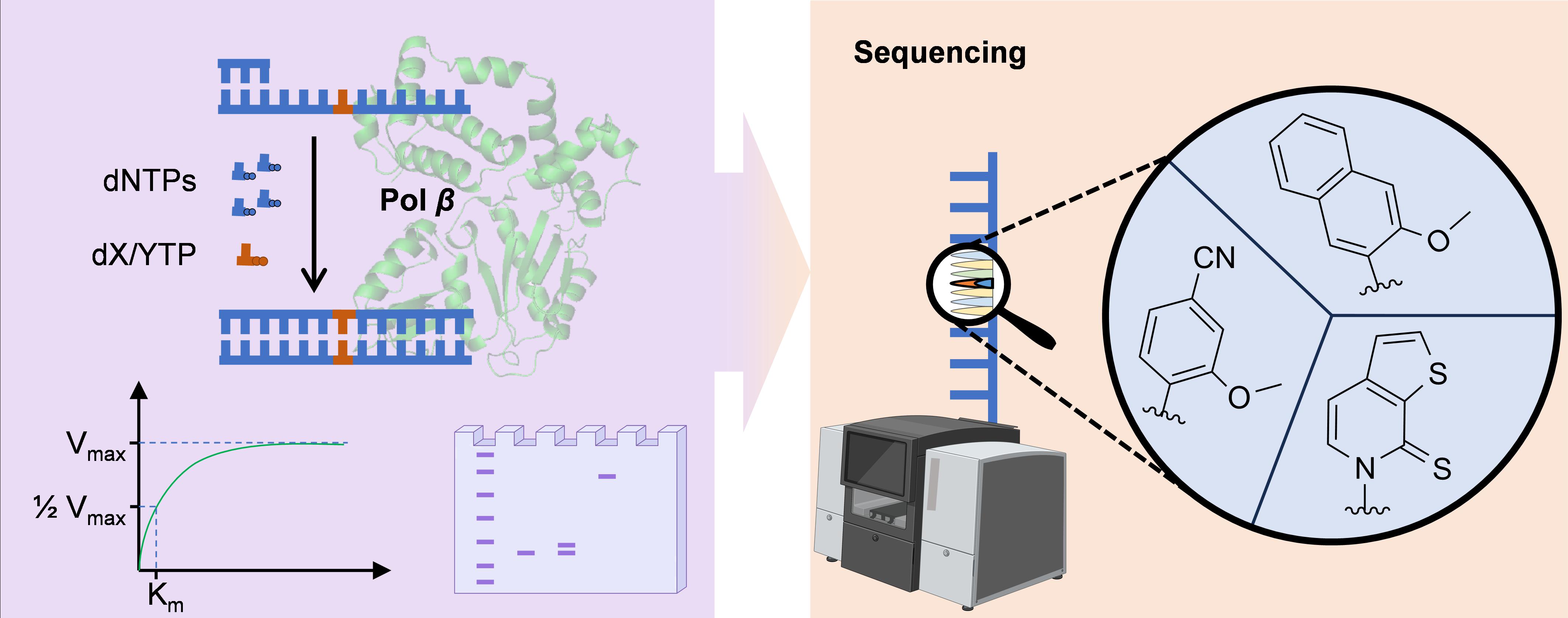
在本工作中,团队首先探索并系统表征了人源DNA聚合酶Pol β对代表性非天然碱基对及其衍生物(dNaM-dTPT3、dNaM-dTPT3Al、dNaM-dTPT3Am、dCNMO-dTPT3、dCNMO-dTPT3Al、dCNMO-dTPT3Am等)的识别、合成、合成后延伸引物的活性及通过这些非天然碱基对的合成进行DNA缺口填补及DNA缺口填补后链置换合成DNA的活性。结果显示,Pol β能够在引物延伸或者DNA缺口填补过程中以较高的效率精准合成包括dNaM-dTPT3、dCNMO-dTPT3等在内的各种代表性非天然碱基对并在非天然碱基对合成后进一步进行引物延伸或链置换DNA合成,展现出优良的催化效率和底物特异性。随后,团队利用Pol β能够严格区分天然与非天然底物的特性,创造性地开发了一套全新的非天然碱基DNA通用测序策略。该方法的核心步骤包括:1)利用Pol β进行引物延伸并在非天然碱基位点前暂停;2)采用Taq聚合酶将非天然碱基选择性替换为两种不同的天然碱基(如dA或dC)并在替换后进一步进行引物延伸;3)通过对引物延伸产物的常规PCR与测序,比对两路测序结果中对应位置不同碱基峰面积的比例差异,从而确定非天然碱基的准确位置。该方法被成功应用于含单个、多个非天然碱基的固定序列,以及含单个非天然碱基的随机序列文库的测序,展现出优良的准确性和普适性。
本研究首次在分子层面证实,真核细胞的DNA复制与修复系统具备识别和处理非天然碱基(对)的潜力,为将来在酵母、动植物细胞乃至更复杂的真核系统中实现遗传字母和遗传密码的扩展、构建真核半合成生命体(eukaryotic semi-synthetic organism,eSSO)铺平了道路。所建立的非天然碱基DNA测序技术操作简便,结果精准,适用于各种含有不同非天然碱基的DNA的测序,且与主流Sanger测序及高通量测序平台直接兼容,为扩展遗传字母的DNA片段提供了稳定、可靠且易于推广的测序方案,为非天然碱基在DNA数据存储、分子进化(如SELEX)及生物医学检测等领域的实际应用奠定了关键的技术基础,具有重要的科学意义与广阔的应用前景。
该研究得到了国家自然科学基金面上项目、广东省“珠江人才计划”引进创新创业团队、广东省“珠江人才计划”青年拔尖人才项目、国家重点研发计划“合成生物学”重点专项、以及广州市科技局基础与应用基础研究项目的资助。