王领副教授课题组开发基于官能团的分子表示学习通用AI大模型
近日,我院王领副教授课题组在生物信息学和计算生物学领的权威期刊Briefings in Bioinformatics上发表题为“FG-BERT: a generalized and self-supervised functional group-based molecular representation learning framework for properties prediction”的研究论文,2021级硕士生李标顺为论文的第一作者,王领副教授为通讯作者。

准确的分子性质预测对于功能分子的设计和发现具有重要意义,特别是对于药物分子的发现,因为它可以在药物发现的早期阶段用于快速识别具有理想性质的活性分子。通常,分子表征是分子性质预测的基础。因此,如何获得有效的分子表征是功能分子性质预测领域需要解决的重要问题。目前的分子表征可分为五类:分子描述符、分子指纹、分子图、分子字符串和分子图像。基于这些预定义的分子表征,研究者可以利用机器学习(ML)和深度学习(DL)来构建用于预测功能分子性质的定量结构-活性/性质关系(QSAR/QSPR)模型。
基于监督学习的ML和DL方法,存在固有缺陷。一方面,传统ML方法很大程度上取决于如何选择合适的分子表示,即分子特征的合理设计与选择。另一方面,监督DL学习方法可以克服传统ML方法的特征设计,但其高度依赖大量且有标签(标记)的数据,而有标记的药物发现数据集往往比较少(即稀缺性)。此外,分子的性质与其结构密切相关,而官能团(FG)是分子中重要的子结构。为此,我们对1456893个分子中的FG种类和数量进行统计(图1)显示:大量FG广泛存在于小分子药物中,侧面表明FG在小分子药物的性质中发挥着至关重要的作用。然而,现有AI药物设计方法很少关注FG的作用。
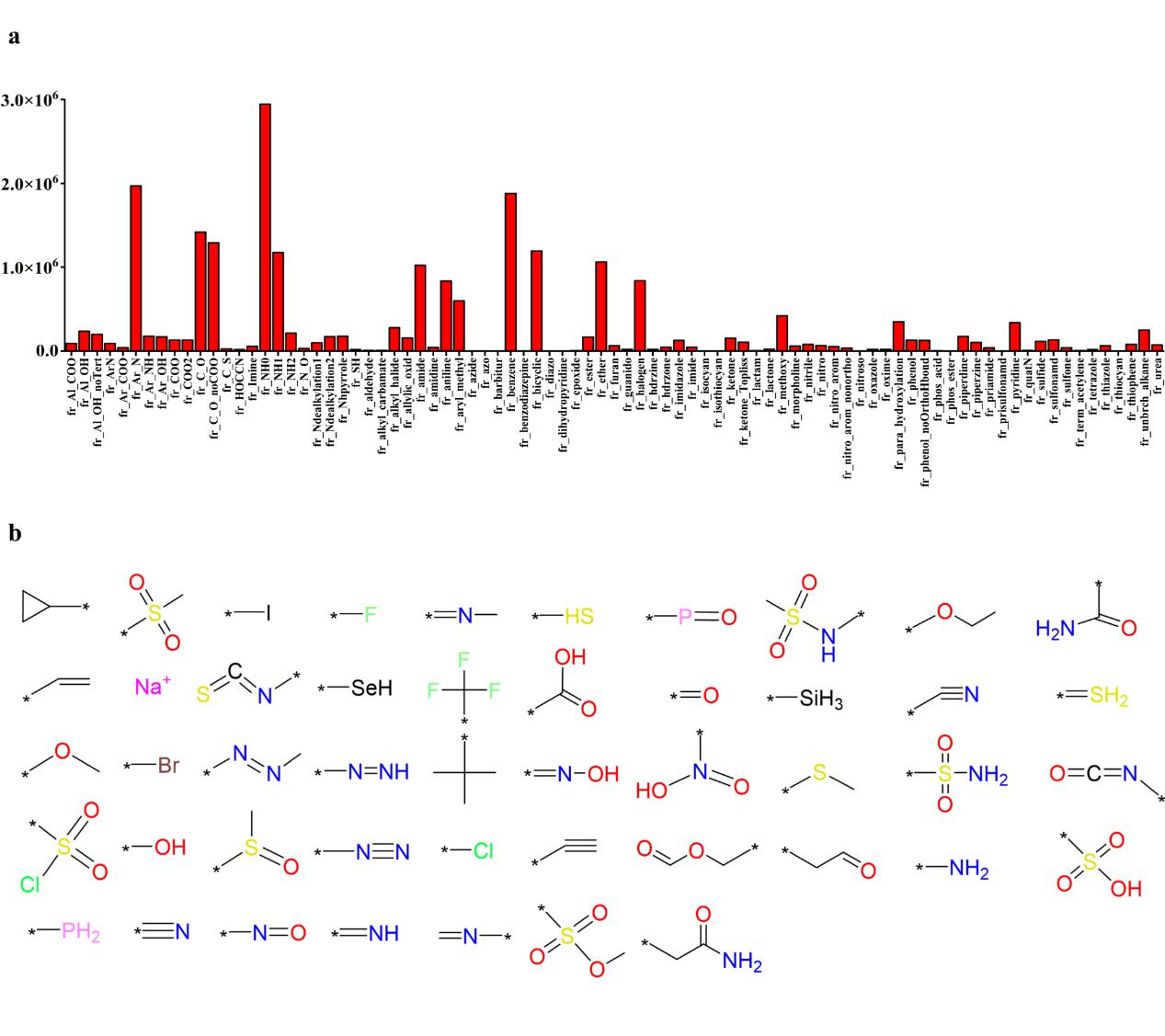
图1. 官能团数量统计
基于上述科学问题,在本研究中,我们开发了一个基于官能团掩蔽-恢复的化学语言预训练通用模型(Functional Group Bidirectional Encoder Representations from Transformers,即FG-BERT,图2),用于从大规模的未标记分子语料库中学习化学语义和结构信息。FG-BERT有两个重要的改进:(1)它将官能团(FG)屏蔽在分子中以执行大规模的高精度预训练恢复预测;(2)它利用一个自我监督的预训练学习框架从∼145万个具有不同生物活性的类药物分子中学习有用的分子表示。
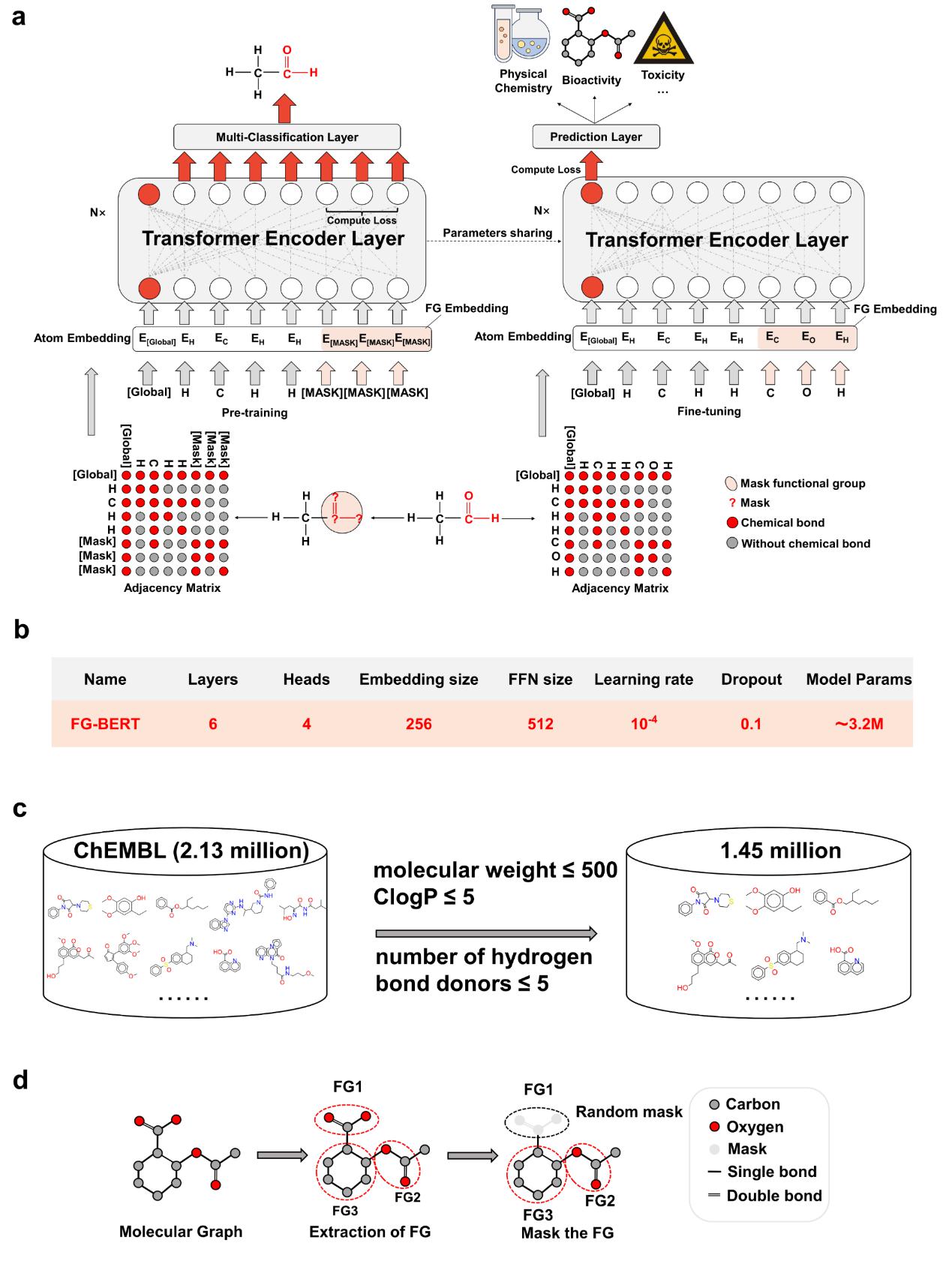
图2. FG-BERT算法的总体框架
作者在涵盖物理化学、生物物理学、生理学性质和毒性的12个药物发现相关的基准数据集上系统地评估了FG-BERT模型的性能。实验表明(表1&表2),与所有基线模型相比,FG-BERT模型在12个基准数据集的学习任务中的8个任务上实现了最先进的性能。此外,详细的消融研究表明(图3),预训练策略和FGs信息都可以提高FG-BERT模型的预测性能。
Methods | Tox21 | ToxCast | Sider | ClinTox | MUV | HIV | BBBP | Bace | Average |
InfoGraph [58](Sun et al., 2020b) | 73.3±0.6 | 61.8±0.4 | 58.7±0.6 | 75.4±4.3 | 74.4±1.8 | 74.2±0.9 | 68.7±0.6 | 74.3±2.6 | 70.10 |
GPT-GNN [59](Hu & others., 2020) | 74.9±0.3 | 62.5±0.4 | 58.1±0.3 | 58.3±5.2 | 75.9±2.3 | 65.2±2.1 | 64.5±1.4 | 77.9±3.2 | 68.45 |
EdgePred [60](Hamilton et al., 2017) | 76.0±0.6 | 64.1±0.6 | 60.4±0.7 | 64.1±3.7 | 75.1±1.2 | 76.3±1.0 | 67.3±2.4 | 77.3±3.5 | 70.08 |
ContextPred [61](Hu et al., 2020) | 73.6±0.3 | 62.6±0.6 | 59.7±1.8 | 74.0±3.4 | 72.5±1.5 | 75.6±1.0 | 70.6±1.5 | 78.8±1.2 | 70.93 |
GraphLoG [62](Xu et al., 2021a) | 75.0±0.6 | 63.4±0.6 | 59.6±1.9 | 75.7±2.4 | 75.5±1.6 | 76.1±0.8 | 68.7±1.6 | 78.6±1.0 | 71.56 |
G-Contextual [63](Rong et al., 2020b) | 75.0±0.6 | 62.8±0.7 | 58.7±1.0 | 60.6±5.2 | 72.1±0.7 | 76.3±1.5 | 69.9±2.1 | 79.3±1.1 | 69.34 |
G-Motif [63](Rong et al., 2020b) | 73.6±0.7 | 62.3±0.6 | 61.0±1.5 | 77.7±2.7 | 73.0±1.8 | 73.8±1.2 | 66.9±3.1 | 73.0±3.3 | 70.16 |
AD-GCL [64](Suresh et al., 2021) | 74.9±0.4 | 63.4±0.7 | 61.5±0.9 | 77.2±2.7 | 76.3±1.4 | 76.7±1.2 | 70.7±0.3 | 76.6±1.5 | 72.16 |
JOAO [65](You et al., 2021) | 74.8±0.6 | 62.8±0.7 | 60.4±1.5 | 66.6±3.1 | 76.6±1.7 | 76.9±0.7 | 66.4±1.0 | 73.2±1.6 | 69.71 |
SimGRACE [66](Xia et al., 2022b) | 74.4±0.3 | 62.6±0.7 | 60.2±0.9 | 75.5±2.0 | 75.4±1.3 | 75.0±0.6 | 71.2±1.1 | 74.9±2.0 | 71.15 |
GraphCL [67](You et al., 2020) | 75.1±0.7 | 63.0±0.4 | 59.8±1.3 | 77.5±3.8 | 76.4±0.4 | 75.1±0.7 | 67.8±2.4 | 74.6±2.1 | 71.16 |
GraphMAE [68](Hou et al., 2022) | 75.2±0.9 | 63.6±0.3 | 60.5±1.2 | 76.5±3.0 | 76.4±2.0 | 76.8±0.6 | 71.2±1.0 | 78.2±1.5 | 72.30 |
3D InfoMax [19](Stärk et al., 2022) | 74.5±0.7 | 63.5±0.8 | 56.8±2.1 | 62.7±3.3 | 76.2±1.4 | 76.1±1.3 | 69.1±1.2 | 78.6±1.9 | 69.69 |
GraphMVP [20](Liu et al., 2022a) | 74.9±0.8 | 63.1±0.2 | 60.2±1.1 | 79.1±2.8 | 77.7±0.6 | 76.0±0.1 | 70.8±0.5 | 79.3±1.5 | 72.64 |
MGSSL [69](Zhang et al., 2021) | 75.2±0.6 | 63.3±0.5 | 61.6±1.0 | 77.1±4.5 | 77.6±0.4 | 75.8±0.4 | 68.8±0.6 | 78.8±0.9 | 72.28 |
AttrMask [61](Hu et al., 2020) | 75.1±0.9 | 63.3±0.6 | 60.5±0.9 | 73.5±4.3 | 75.8±1.0 | 75.3±1.5 | 65.2±1.4 | 77.8±1.8 | 70.81 |
Mole-BERT [21](Xia et al., 2023) | 76.8±0.5 | 64.3±0.2 | 62.8±1.1 | 78.9±3.0 | 78.6±1.8 | 78.2±0.8 | 71.9±1.6 | 80.8±1.4 | 74.04 |
FG-BERT | 78.4±0.8 | 66.3±0.8 | 64.0±0.7 | 83.2±1.6 | 75.3±2.4 | 77.4±1.0 | 70.2±0.9 | 84.5±1.5 | 74.92 |
表1.FG-BERT算法在分类任务据集上的性能结果
表2. FG-BERT算法在回归任务据集上的性能结果
Methods | ESOL | Lipo | Malaria | CEP | Average |
ContextPred [61](Hu et al., 2020) | 1.196±0.037 | 0.702±0.020 | 1.101±0.015 | 1.243±0.025 | 1.061 |
JOAO [65](You et al., 2021) | 1.120±0.019 | 0.708±0.007 | 1.145±0.010 | 1.293±0.003 | 1.067 |
GraphMVP [20](Liu et al., 2022a) | 1.064±0.045 | 0.691±0.013 | 1.106±0.013 | 1.228±0.001 | 1.022 |
AttrMask [61](Hu et al., 2020) | 1.112±0.048 | 0.730±0.004 | 1.119±0.014 | 1.256±0.000 | 1.054 |
Mole-BERT [21](Xia et al., 2023) | 1.015±0.030 | 0.676±0.017 | 1.074±0.009 | 1.232±0.009 | 0.999 |
FG-BERT | 0.944±0.025 | 0.655±0.009 | 1.057±0.006 | 1.051±0.029 | 0.927 |
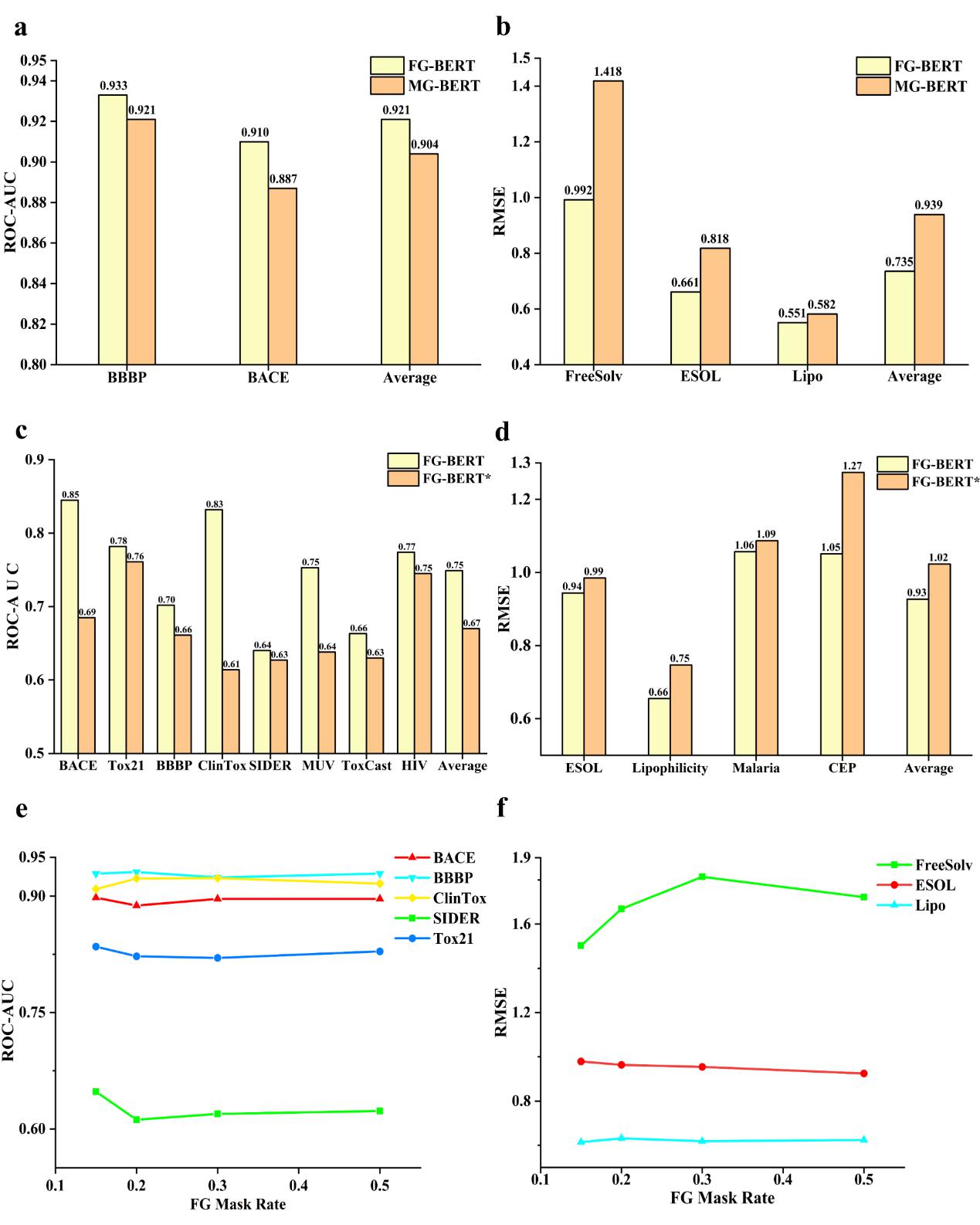
图3. FG-BERT的消融实验研究结果
此外,作者还在15个ADMET数据集和14个基于细胞的表型筛选数据集上进一步对FG-BERT预训练模型性能进行了评估,结果如图4所示,FG-BERT模型的预测性能均达到最优,其平均AUC分别为0.813和0.856。这些结果充分表明FG-BERT是预测分子 ADMET 性质最具竞争力的方法之一,并且在基于细胞的表型筛选药物发现方面具有巨大的潜力。
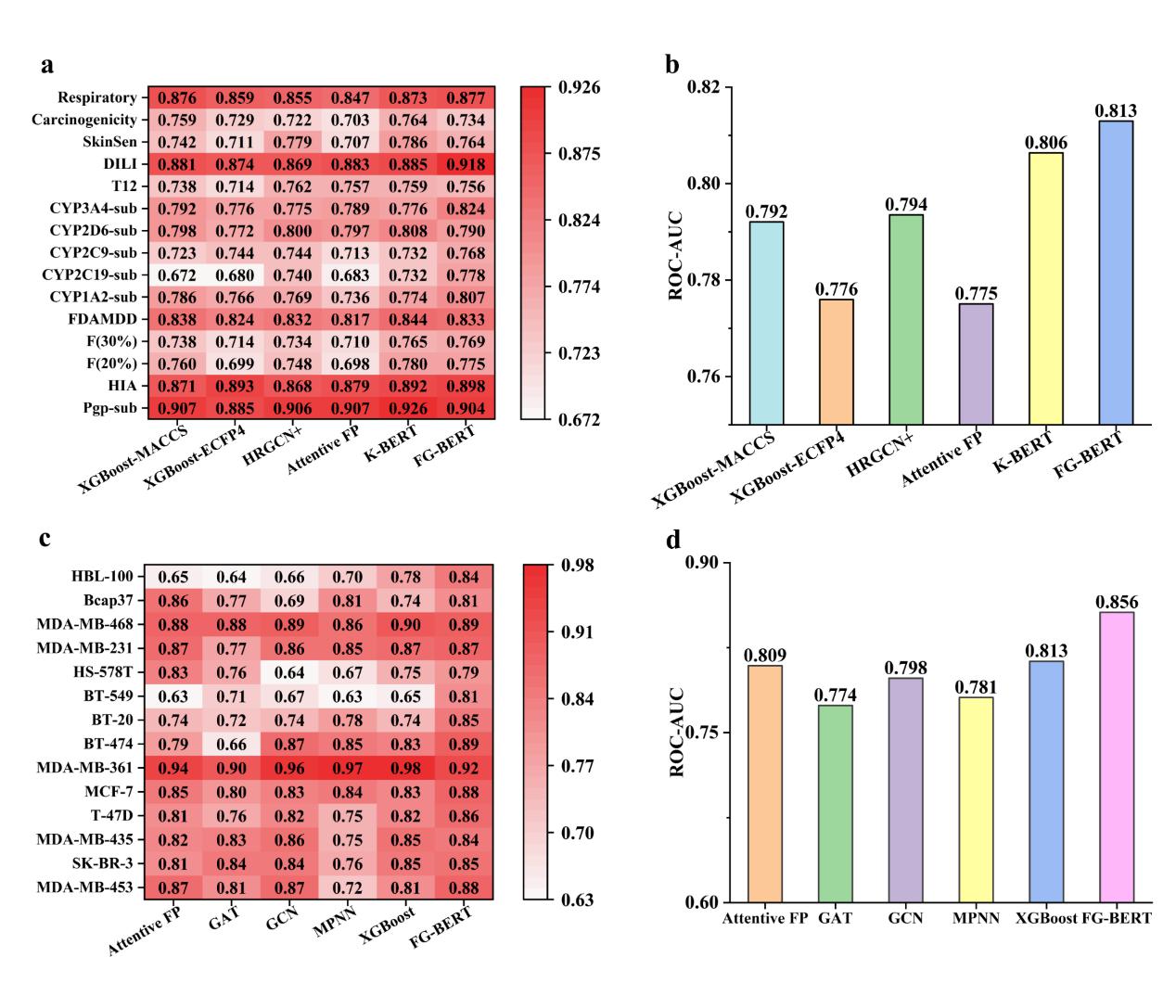
图4. FG-BERT在15个ADMET数据集和14个细胞表型筛选数据集的预测结果
最后,在案例分析中(图5),作者通过将注意力机制可视化来验证了FG-BERT模型的可解释性。通过对FGs进行掩蔽-恢复预训练,FG-BERT能识别出BACE以及BBBP数据集中分子的关键成分,从而为化学家提供了药物性质预测中具有重要价值的官能团/分子片段。
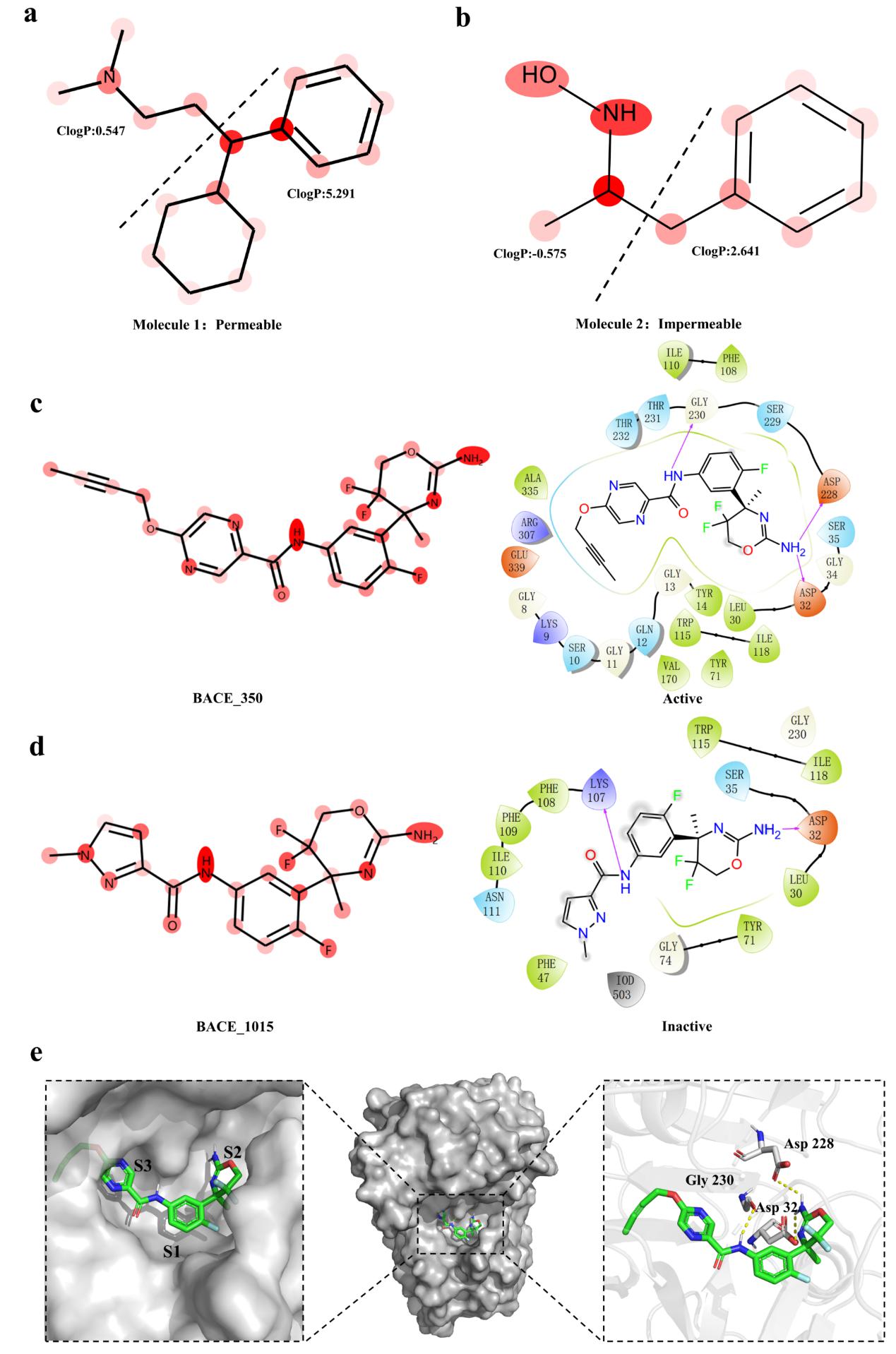
图5. 模型在BACE和BBBP数据集上的可解释性分析
总而言之,FG-BERT 作为一种开箱即用、有效且可解释的计算工具,可用于各种与药物发现相关的任务。作者期望FG-BERT作为强大的深度学习工具,可以帮助化学家识别分子中的关键结构,从而设计出具有预期特性或功能的分子。
上述研究结果得到国家自然科学基金面上项目(81973241)和广东省自然科学基金杰出青年项目(2023B1515020042)的支持。
原文链接:https://academic.oup.com/bib/article-abstract/24/6/bbad398/7337693?redirectedFrom=fulltext
代码链接:https://github.com/idrugLab/FG-BERT