陈庭坚教授团队在ACS Synthetic Biology上发表论文——基于非天然核酸(XNA)聚合酶定向进化的中心法则高维拓展
近日,华南理工大学生物科学与工程学院陈庭坚教授课题组在非天然核酸聚合酶(XNA聚合酶)的分子改造及其在中心法则高维拓展中的应用研究中取得新进展,相关成果发表在期刊ACS Synthetic Biology上,题为:Synthesis, Reverse Transcription, Replication, and Inter-Transcription of 2’-Modified Nucleic Acids with Evolved Thermophilic Polymerases: Efforts toward Multidimensional Expansion of the Central Dogma,硕士生覃彦嘉为论文的第一作者,博士生马星雲、硕士生陶睿、副研究员杜宇辉为共同作者,陈庭坚教授为通讯作者,华南理工大学生物科学与工程学院为唯一单位。(论文链接:https://doi.org/10.1021/acssynbio.3c00213)
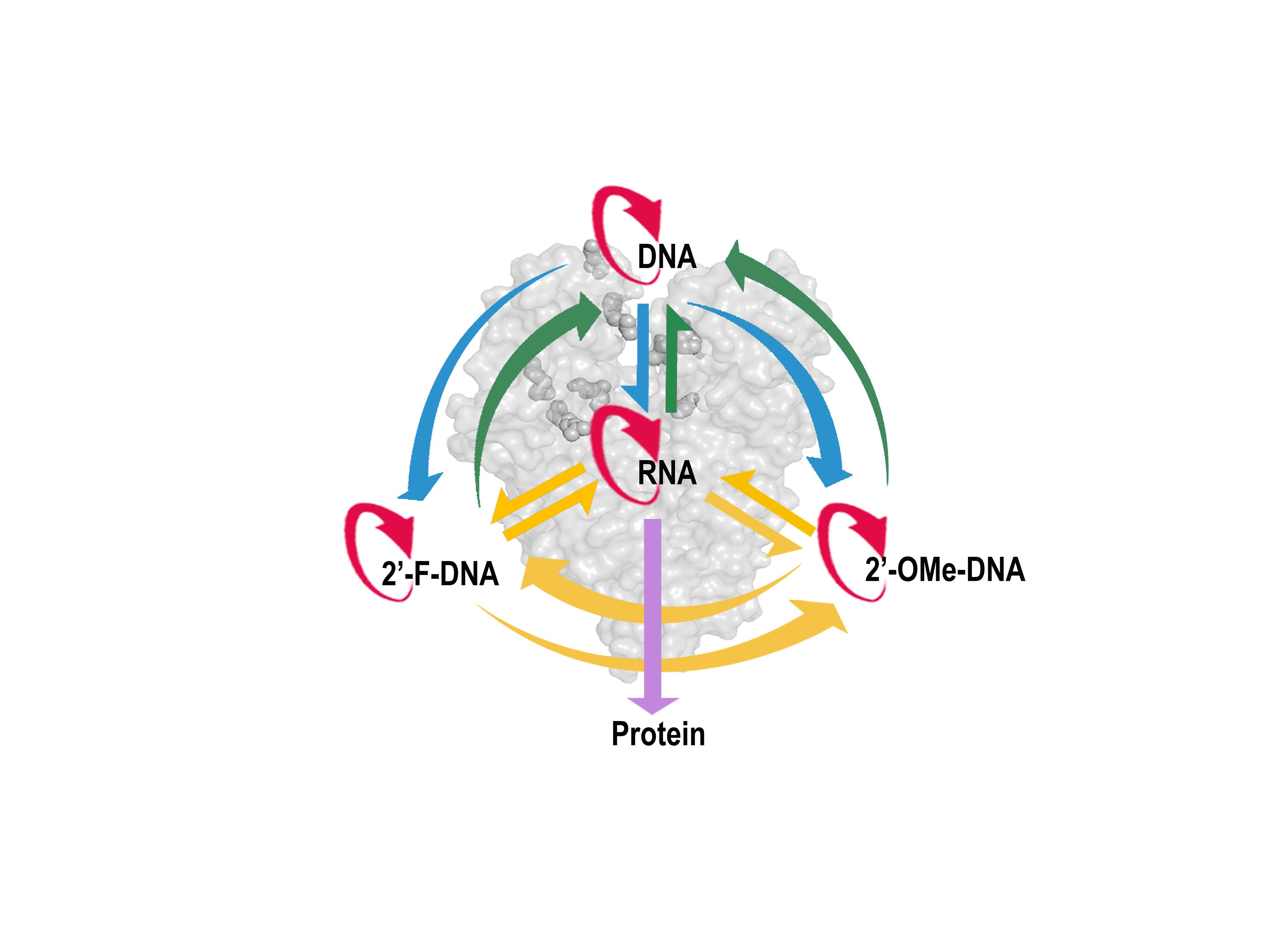
图1. 基于非天然核酸及非天然核酸聚合酶的中心法则高维拓展
包括2’-修饰核酸在内的非天然核酸(xenobiotic nucleic acid,xeno-nucleic acid,XNA,或称异种核酸)作为新型的遗传信息存储介质,在合成生物学、生物技术和生物医药等诸多领域展现出了巨大的应用潜力。要实现XNA的充分利用,拥有能够高效合成、 转录、逆转录甚至扩增XNA的非天然核酸聚合酶十分必要。针对以上问题,前期工作已经对来自不同家族的DNA聚合酶和RNA聚合酶进行了分子改造,获得了一系列能够进行全修饰XNA合成和逆转录以及部分修饰XNA扩增的非天然核酸聚合酶。然而,这些聚合酶合成大长度XNA片段的能力还有待提高,同时复制XNA和进行不同XNA之间相互转录的能力仍需要探索或者建立。
在本工作中,课题组在结构分析的基础之上,对源自Taq DNA聚合酶Stoffel片段的非天然核酸聚合酶SFM4-3手指和拇指结构域表面的关键残基E520和K681进行定点饱和突变建立分子文库,接着通过对该文库进行基于XNA合成活性的高通量筛选,获得了两个2’-OMe转录活性显著增强的突变株SFM5-7和SFM5-46。进一步的实验表明,这两个突变株能够高效转录大长度全修饰的2’-OMe-DNA片段,并且具有良好的保真度(图2)。
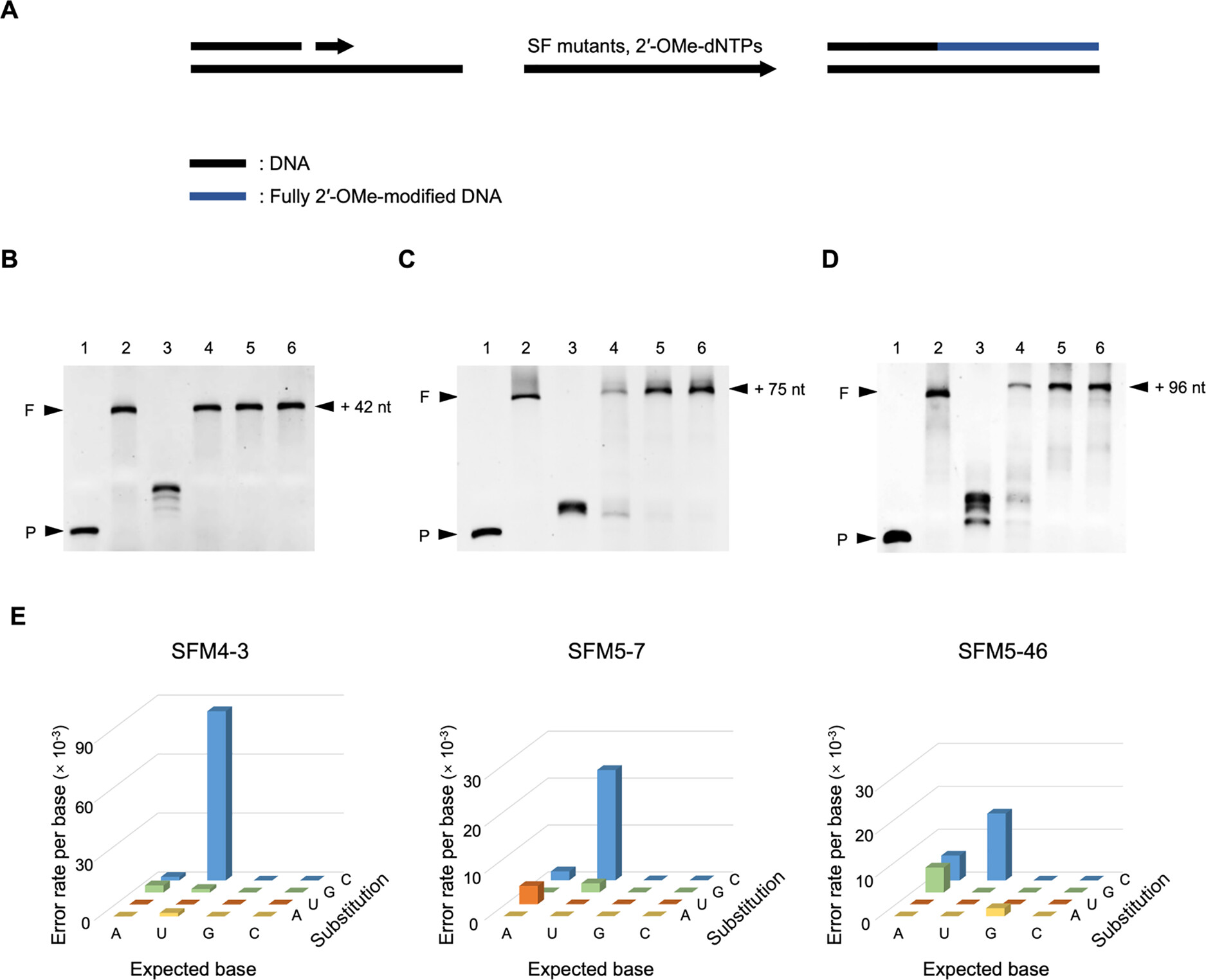
图2. 不同SF突变株转录全修饰2’-OMe-DNA的活性比较和保真度测试
对所得SF突变株进行突变位点分布的分析发现,SFM5-7和SFM5-46的520位点均由谷氨酸突变为脯氨酸,而681位点则由赖氨酸分别突变为精氨酸和亮氨酸,这些突变可能导致了手指和拇指结构域之间相互作用的改变,有利于形成更加稳定的封闭构象;同时SFM5-46的520位点之后额外插入了一段包含18个氨基酸的序列,其中包含的3个精氨酸很可能增强了聚合酶与模板/引物之间的静电相互作用,从而提高了SFM5-46的活性(图3)。
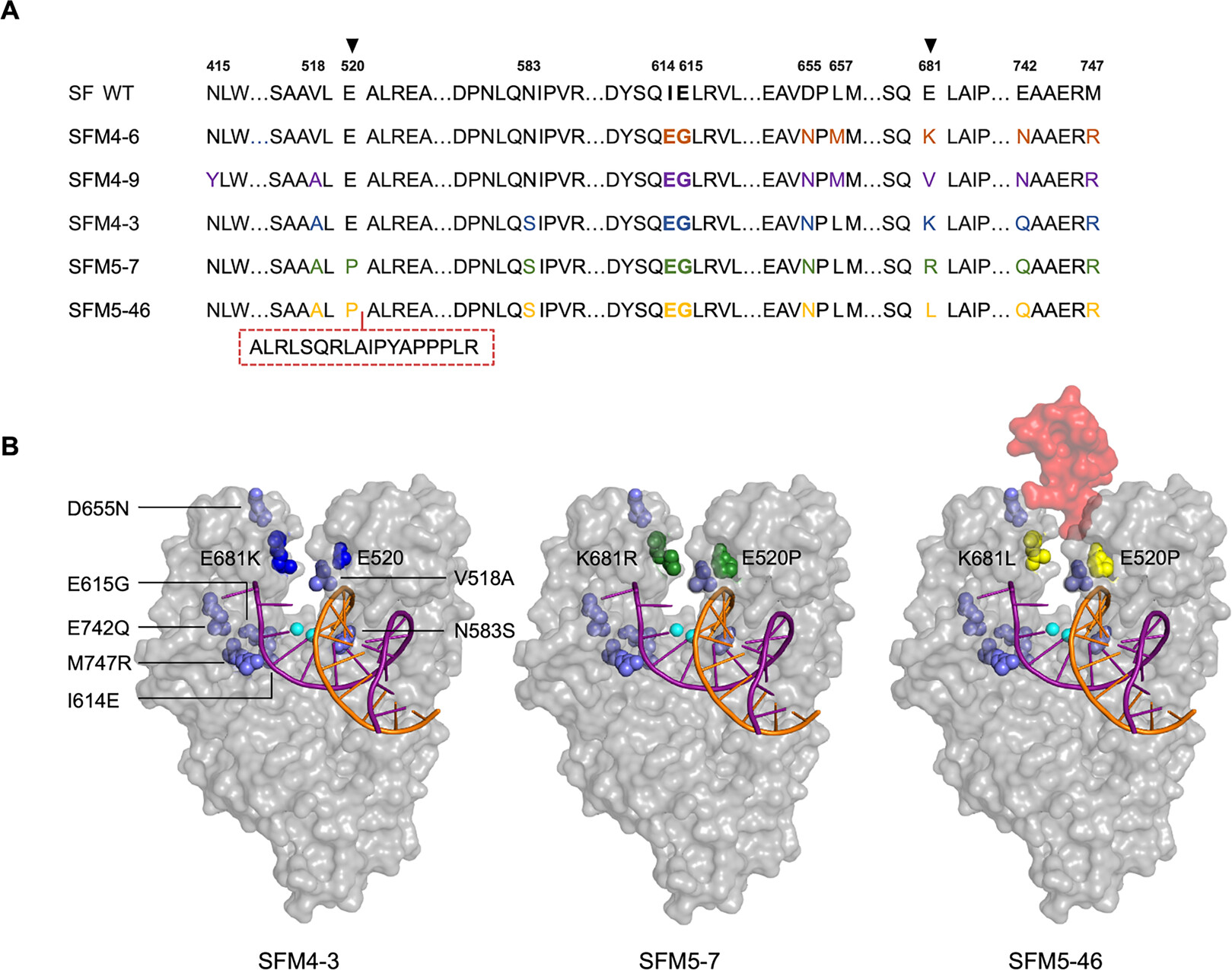
图3. SF突变株的突变位点分布
课题组进一步对所得突变株的其他非天然活性进行了测试和表征。首先,课题组测试了SF突变株以2’-F-DNA为模板合成不同核酸分子的活性。结果显示,SFM4-3、SFM5-7和SFM5-46三个突变株均能以2’-F-DNA为模板高效合成DNA、2’-F-DNA、RNA和2’-OMe-DNA(图4)。
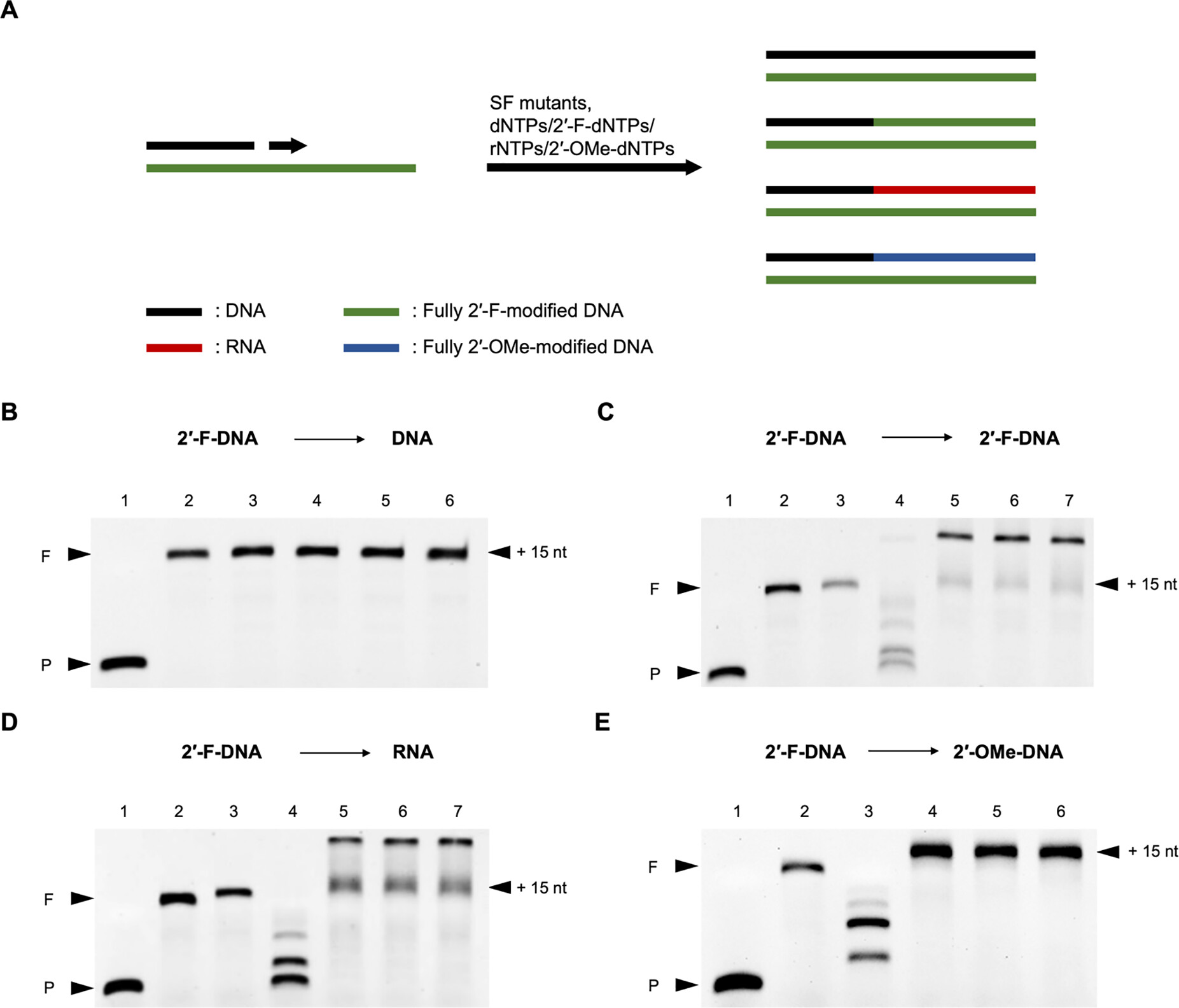
图4. 以2’-F-DNA为模板合成不同核酸分子的活性测试
随后,课题组测试了三个突变株以RNA为模板合成不同核酸分子的活性。结果显示,三个突变株均能以RNA为模板高效合成DNA、2’-F-DNA、RNA,但以 RNA为模板合成2’-OMe-DNA的活性还有待进一步提高(图5)。
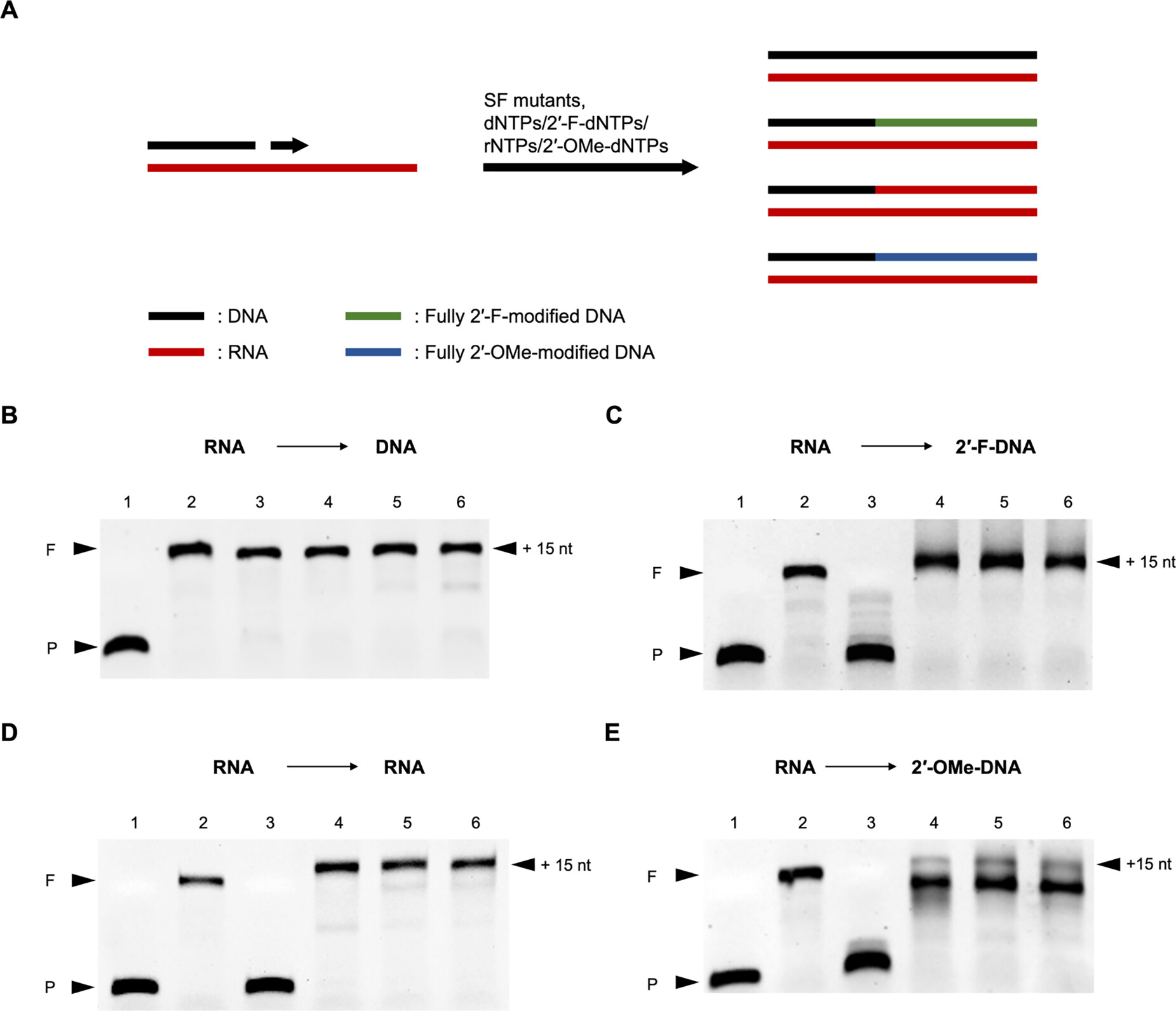
图5. 以RNA为模板合成不同核酸分子的活性测试
最后,课题组测试了三个突变株以2’-OMe-DNA为模板合成不同核酸分子的活性。结果表明,使用SF的突变株可以实现以2’-OMe-DNA为模板的DNA、2’-F-DNA、RNA和2’-OMe-DNA合成。在2’-OMe-DNA的反转录和以2’-OMe-DNA为模板的2’-F-DNA合成中,SFM5-7、SFM5-46的活性优于SFM4-3;而在2’-OMe-DNA复制中,SFM5-7的活性明显优于SFM4-3和SFM5-46(图6)。
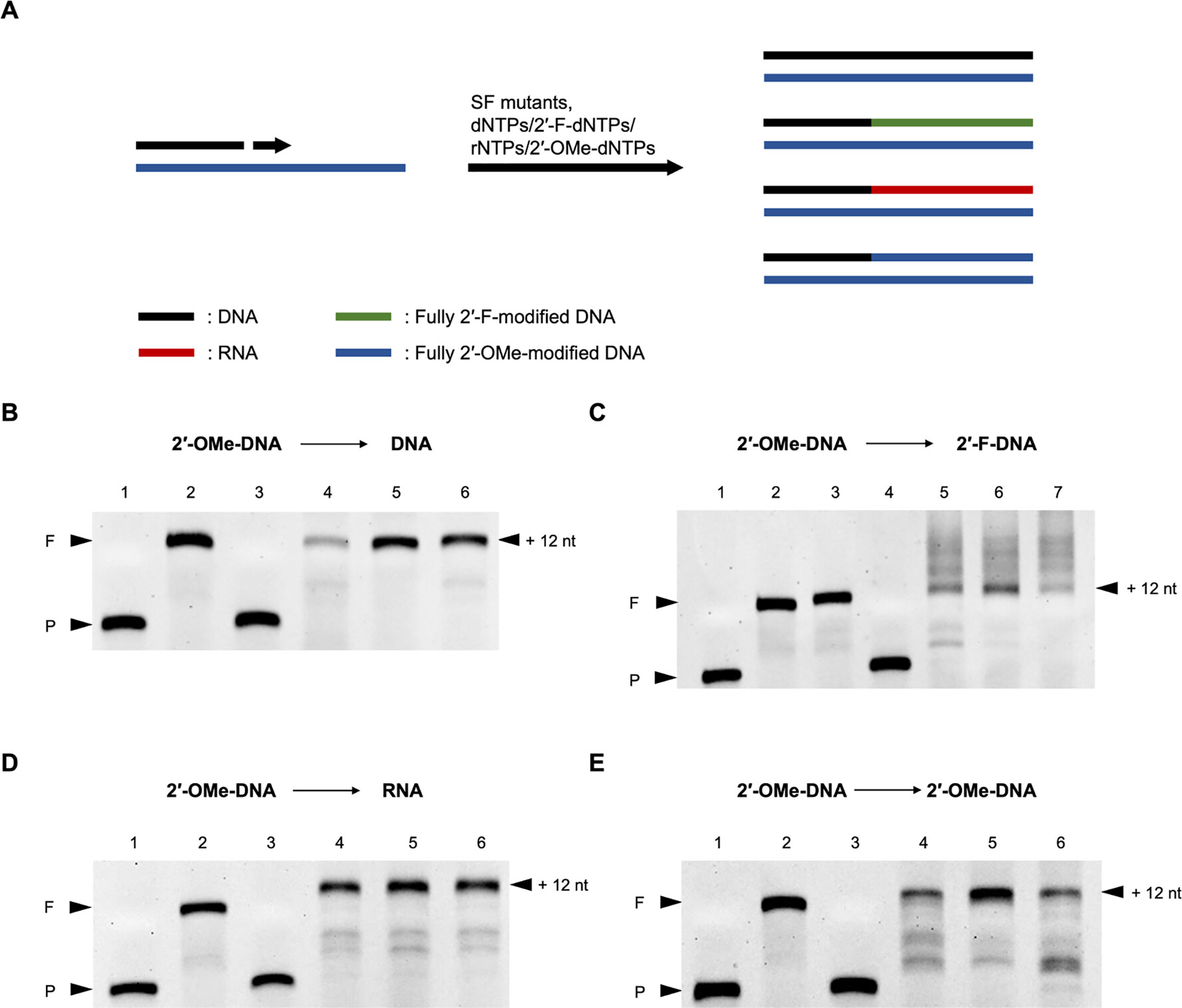
图6. 以2’-OMe-DNA为模板合成不同核酸分子的活性测试
利用非天然核酸聚合酶SFM5-7的2’-OMe-DNA复制活性和以2’-OMe-DNA为模板合成2’-F-DNA的活性,课题组成功建立了快速制备两端由XNA同源双链(2’-OMe-DNA)或异源双链(2’-OMe-DNA/2’-F-DNA)保护的线性DNA的方法,显著提高了线性DNA对核酸外切酶的耐受性(图7)。该策略可以用于方便快速地制备具有优良生理稳定性的末端保护线性DNA模版,有望在无细胞转录和翻译、基因重组和基因编辑等领域获得广泛应用。
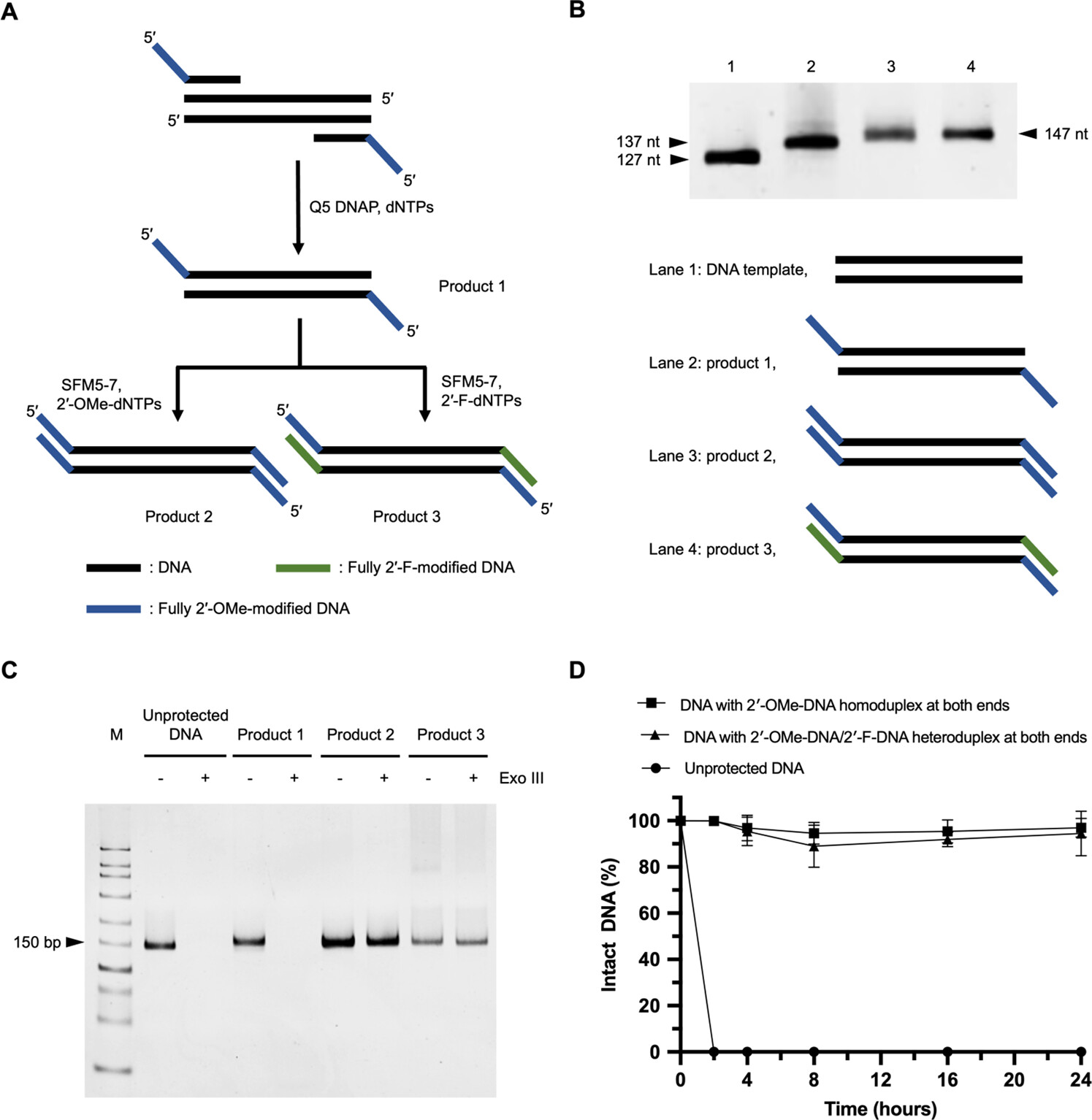
图7. 利用非天然核酸聚合酶突变株SFM5-7酶法制备两端由双链XNA保护、具有优良生理稳定性的线性DNA
综上,本研究通过半理性改造策略,成功获得了能够高效合成大长度全修饰非天然核酸2’-OMe-DNA的SF突变株SFM5-7和SFM5-46。这些非天然核酸聚合酶突变株合成大长度非天然核酸(XNA)的能力,使制备或获取具有较大分子量的非天然核酸适体(XNA适体)、非天然核酸酶(XNA酶,XNAzyme)、非天然核酸材料(如XNA框架、XNA折纸和XNA水凝胶等)构筑基元和其它功能性的非天然核酸分子(如可用于基因编辑的gXNA)成为可能。同时,通过使用这些非天然核酸聚合酶突变株,建立了XNA和RNA的转录、逆转录、复制和互转录(inter-transcription)体系,实现了遗传信息在DNA、RNA、2’-F-DNA和2’-OMe-DNA的任意两种分子之间的自由流动,展示了XNA作为新型遗传信息载体、遗传信息信使/中继站和功能执行分子的潜力,从而将中心法则拓展到了包含各种XNA节点在内的更高维度。该研究全方位展示了利用DNA聚合酶定向进化获取的非天然核酸聚合酶(XNA聚合酶)在非天然核酸(XNA)中写入、复制、扩增和读取信息的可行性,为全面开发非天然核酸(XNA)信息存储技术体系奠定了基础,对开发基于多类型遗传信息分子的稳定信息存储介质和新型可加密高保真信息存储技术具有重要意义。
该工作得到了国家自然科学基金面上项目(21978100),国家重点研发计划“合成生物学”重点专项(2019YFA0904102),广东省“珠江人才计划”青年拔尖人才项目(2019QN01Y228),以及广东省“珠江人才计划”引进创新创业团队(2019ZT08Y318)的资助。