王领副教授课题组开发FP-GNN深度学习算法用于分子性质的精准预测
近年来,人工智能辅助分子性质(如理化性质,药理活性,ADMET等)预测成为药物开发领域的研究热点。然而人工智能辅助分子性质预测领域尚存在许多难题,例如现有的机器学习/深度学习算法准确度还有待提高,生物相关数据集中噪声数据多,子任务之间可能存在着紧密联系以及深度学习算法难以解释等问题。那么,探索一种具有更全面分子表征方式、更优秀的预测性能、更好的抗噪声能力和直观的可解释性的深度学习算法在药物研发领域具有重要意义。
近日,我院王领副教授课题组在Briefings in Bioinformatics(IF=14)上发表了题为FP-GNN: a versatile deep learning architecture for enhanced molecular property prediction的论文。基于上述人工智能辅助分子性质预测领域存在的问题,本文首次提出并开发了一种基于分子图和分子指纹相融合的深度学习分子性质预测算法,名为FP-GNN(Fingerprint and Graph Neural Networks)。FP-GNN深度学习算法总体分为特征提取的分子图模块(Graph Neural Networks, GNN)、分子指纹信息模块(Fingerprint Networks, FPN),和特征融合输出的综合模块(Fully Connected Networks, FCN)。如图1所示,FP-GNN模型收到分子的SMILES表征后,将其处理成分子图结构数据和分子指纹,分别输入GNN和FPN模块。GNN从分子图中自动提取分子的理化和结构特征;FPN从分子指纹中提取化学家们预先定义好的先验理化信息和结构信息;FCN将GNN和FPN模块中得到的特征进行融合,并做最优信息比例自适配,最后输出模型对分子性质的预测结果。首先,在三个主流算法测试数据集上,包括机器学习基准数据集MoleculeNet,药物虚拟筛选基准数据集LIT-PCBA以及14个乳腺癌细胞系药物筛选模型上,相比传统机器学习如Random Forest(RF)、Support Vector Machine(SVM)、Naive Bayesian(NB)和eXtreme Gradient Boosting(XGBoost)和主流深度学习算法如GCN, MPNN, GAT, D-MPNN,Attentive FP和HRGCN+,FP-GNN取得总体最优的预测性能。其次,抗干扰测试证明了FP-GNN模型具有优秀的抗干扰能力,说明FP-GNN模型能够适用于真实场景中,促进药物研发领域的实际开发过程。最后,FP-GNN模型还具有可解释性,包括分子图模块和分子指纹模块的双重解释性,提高模型的信服力,为使用者提供了药物性质预测中具有重要价值的分子片段。2019级硕士生蔡涵萱(现为海康威视(上海)任AI算法开发工程师)为论文的第一作者,王领副教授为通讯作者。
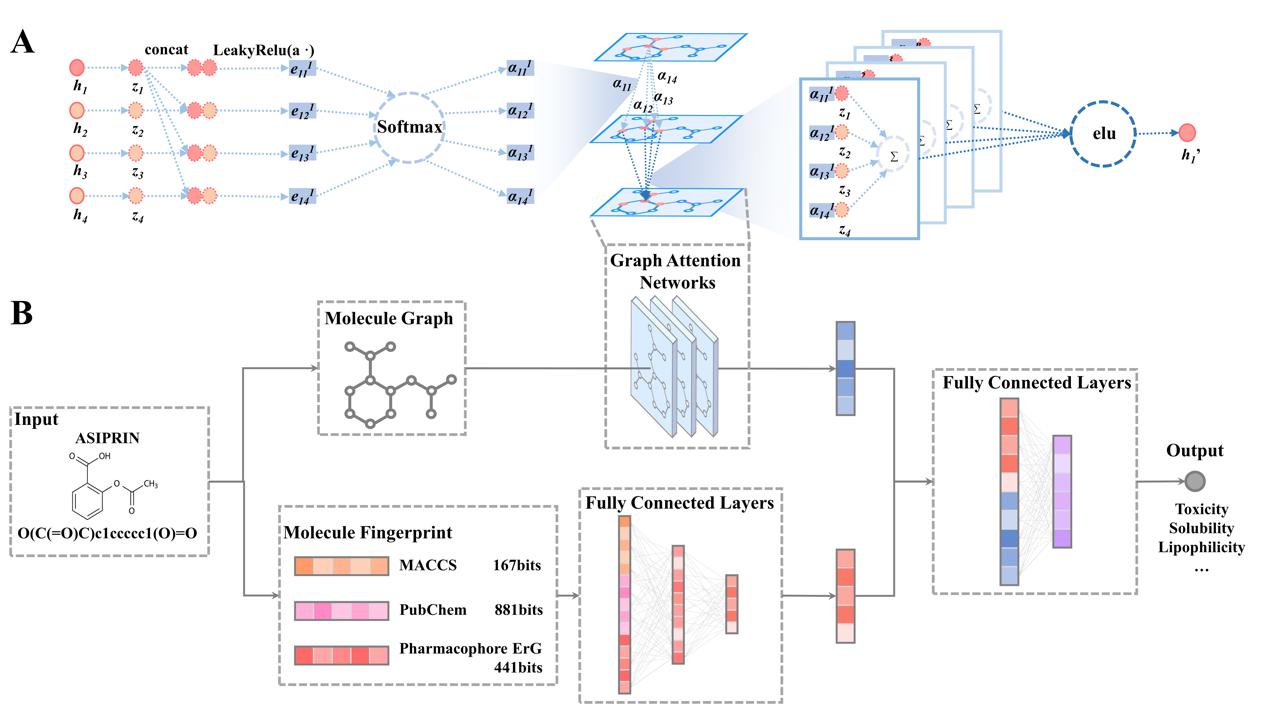
图 1 FP-GNN模型的总体框架
目前,本算法已在课题组内多项药物发现相关课题中投入使用。例如,2019级硕士生张慧敏研究使用FP-GNN模型在细胞周期依赖性家族激酶9(CDK9)上进行建模,成功筛选出了五个靶向抑制CDK9的化合物,并通过体外细胞实验证明了这五个化合物在八种肿瘤细胞上具有较好的抗癌活性。其中化合物C9在MOLM-13细胞(急性髓系白血病细胞株)中显示出较强的细胞抑制活性(IC50 = 3.92 μM)和CDK9抑制活性(IC50 = 295 nM)。如图2所示,作用机制研究结果表明化合物C9可抑制MOLM-13细胞内CDK9的活性,并诱导细胞凋亡的产生。研究结果在Bioorganic & Medicinal Chemistry(IF=3.459)上发表(Ligand- and structure-based identification of novel CDK9 inhibitors for the potential treatment of leukemia )。2019级硕士生张慧敏(现为深圳晶泰科技新药发现部助理研究员)为论文的第一作者,王领副教授为通讯作者。
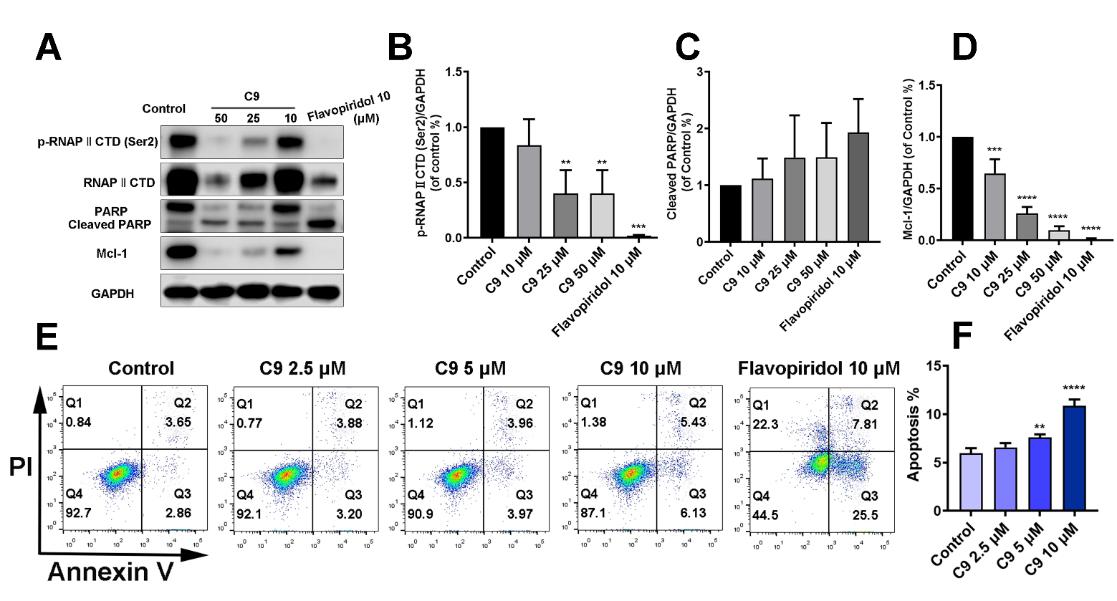
图2 化合物C9对CDK9通路蛋白和细胞凋亡的影响
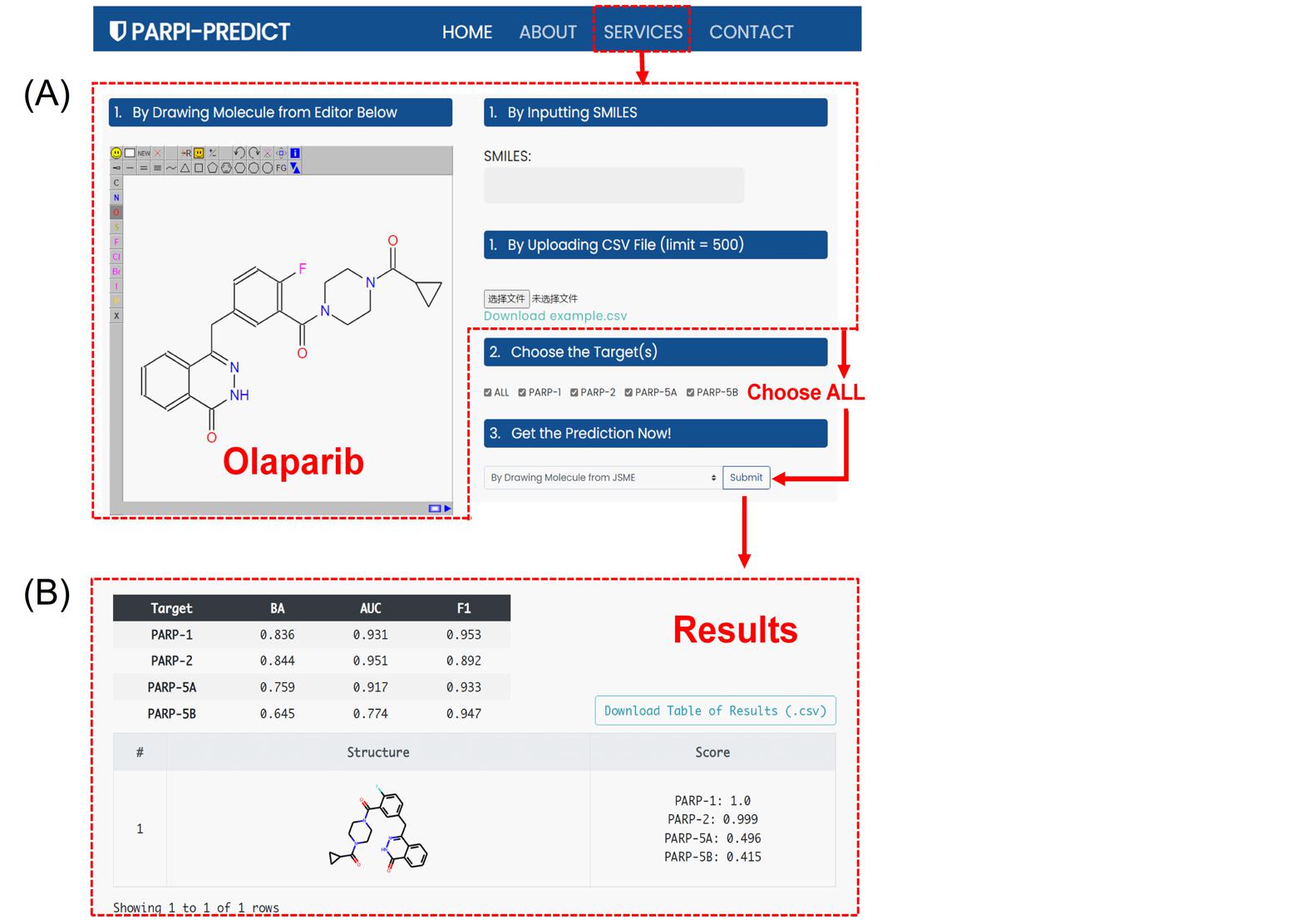
图3 PARPi-Predict平台预测功能展示
最后,考虑到药物发现领域中,许多数据集的子任务之间往往存在着较强的相关性,如果只采用单任务模型进行训练预测,会丢失子任务之间的数据关联信息。课题组对FP-GNN算法进行多任务建模框架的开发,并对多任务模型进行了深入的评估分析。研究发现多任务FP-GNN模型应用于数据相关性强的聚腺苷二磷酸核糖聚合酶家族(PARPs)上,可以捕捉子任务间的相关信息,显著提升模型的预测性能。为此课题组开发了基于多任务FP-GNN模型的新型PARP选择性抑制剂设计与预测平台(PARPi-Predict:https://parpipredict.idruglab.cn,图3),以方便该领域的研究者设计和发现新型选择性PARP抑制剂用于治疗癌症等疾病。研究结果在Frontiers in Pharmacology(IF=6)上发表(A multi-task FP-GNN framework enables accurate prediction of selective PARP inhibitors)。2020级硕士生艾带巧和2019级生物制药班本科生巫景行为并列第一作者,王领副教授为通讯作者。
综合而言,相比当前优秀的机器学习和深度学习算法,本课题组开发的FP-GNN深度学习算法具有更优秀的预测性能,在药物研发实战中,FP-GNN也能大展身手,加速药物筛选的周期,降低药物筛选的成本,在药物发现领域有着强大的实用价值。FP-GNN算法的源代码和使用说明可以在开源网站上下载得到:http://github.com/idrugLab/FP-GNN。
上述研究结果得到国家自然科学基金面上项目(81973241)和广东省自然科学基金面上项目(2020A1515010548)的支持。
原文链接:
https://academic.oup.com/bib/advance-article-abstract/doi/10.1093/bib/bbac408/6702671?redirectedFrom=fulltext