杜红丽教授课题组在Computational and Structural Biotechnology Journal杂志上在线发表论文:病毒关键候选突变位点和流行趋势自动化分析工具
近日,华南理工大学生物科学与工程学院杜红丽教授课题组在Computational and Structural Biotechnology Journal杂志上在线发表题为AutoVEM2: a flexible automated tool to analyze candidate key mutations and epidemic trends for virus的研究论文。生物科学与工程学院2021届本科毕业生席彬彬为论文第一作者,博士研究生陈子曦、本科生李淑华为论文共同第一作者,杜红丽教授为通讯作者(论文链接:https://doi.org/10.1016/j.csbj.2021.09.002)。
AutoVEM2是在先前课题组开发的针对新冠病毒关键突变位点和突变亚型流行趋势分析工具AutoVEM(Xi, et al. Comp. Struct. Biotechnol. J., 19(2021): 1976-1985)的基础上开发的,旨在为广大研究者提供一个可以自动化分析任何病毒(在提供参考基因组的前提下)关键突变位点和突变亚型流行趋势的工具(图一)。AutoVEM2除扩展了工具的应用范围之外,还将分析流程分成了三个模块并提供了更多的选择参数,研究者可以根据自己的需求进行个性化分析。最后,研究还将开发的工具AutoVEM2应用在了HBV和HPV-16关键突变位点筛选以及特定时期内SARS-CoV-2关键突变位点和亚型流行趋势分析上(图二)。
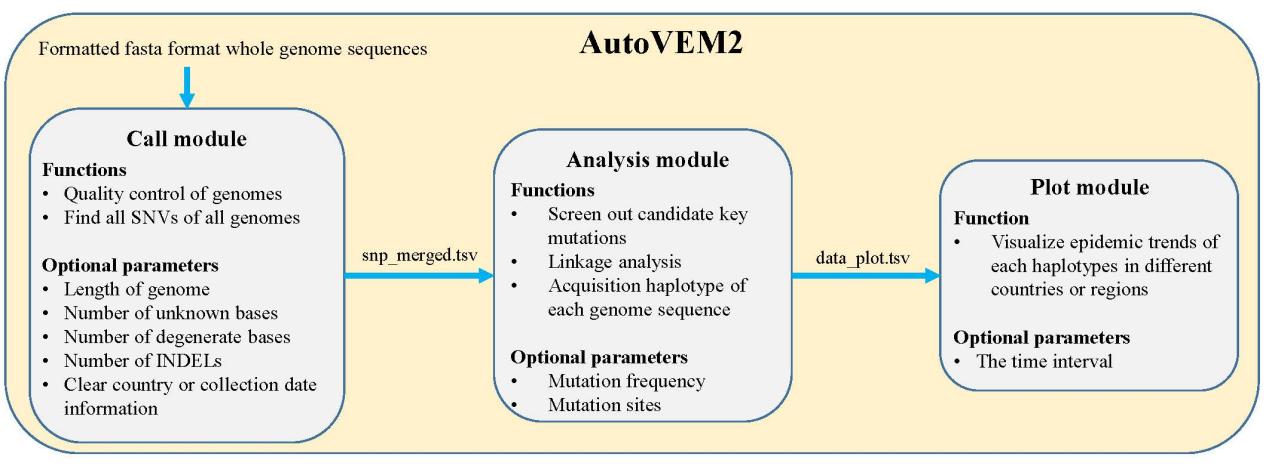
图一:AutoVEM2主要模块和各模块功能
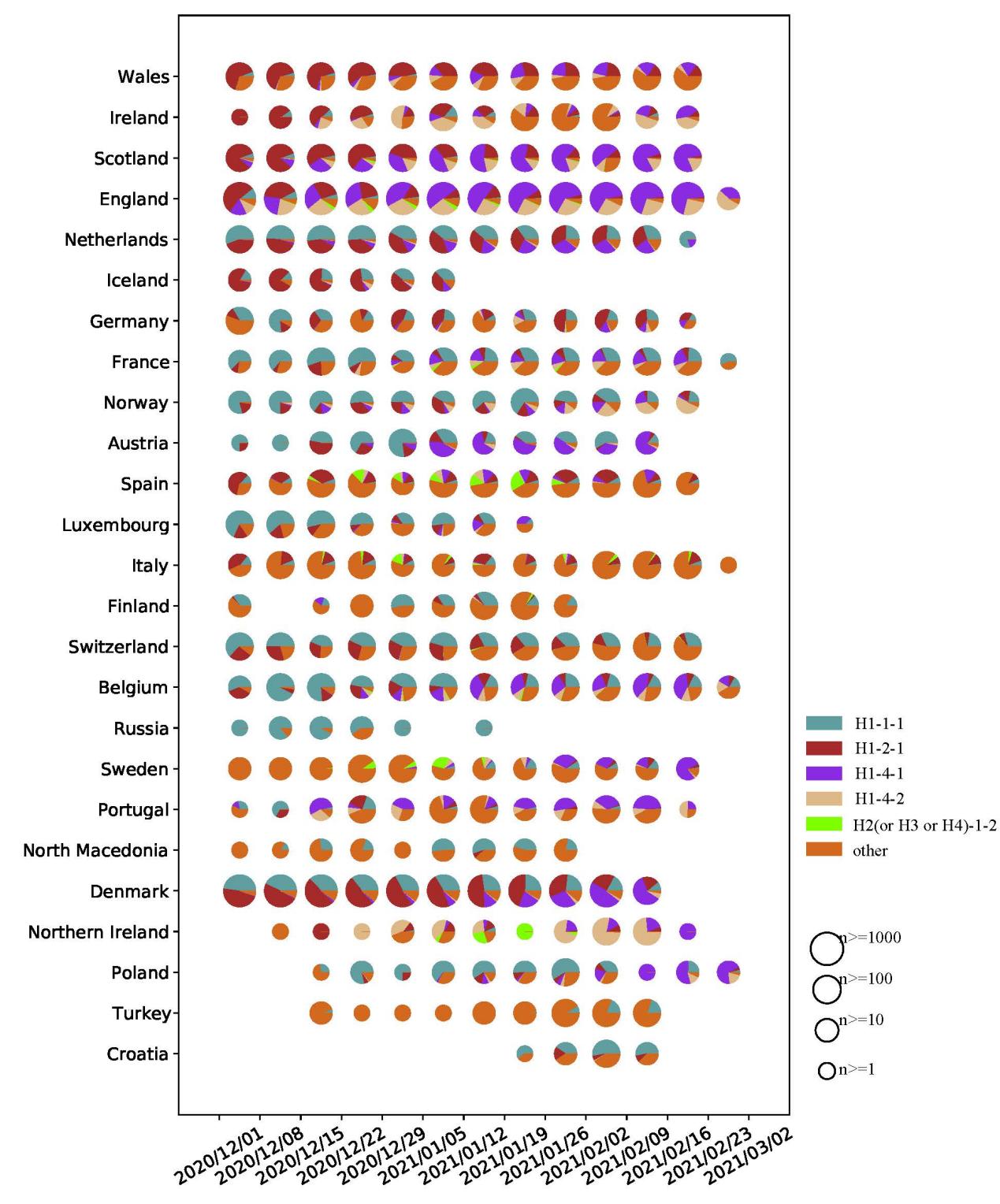
图二:SARS-CoV-2 主要单倍型在欧洲流行趋势
据悉,杜红丽教授课题组今年8月份还在Computational and Structural Biotechnology Journal发表了另外一篇整合组学分析工具ARMT: An automatic RNA-seq data mining tool based on comprehensive and integrative analysis in cancer research(https://doi.org/10.1016/j.csbj.2021.08.009),论文第一作者为博士研究生黄管大。课题组希望这些研究能为广大科研工作者提供更加标准化、高效、易用的分析工具。
该项工作得到国家重点研发计划(2018YFC0910201)和广东省重点研发计划(2019B020226001)等项目资助。